交叉信息研究院曾坚阳研究组2019年发表于《自然·通讯》杂志的研究成果《基于FISH和Hi-C数据整合的三维基因组结构建模》(Integrating Hi-C and FISH data for modeling of the 3D organization of chromosomes),近日经《基因组蛋白质组与生物信息学报》(Genomics, Proteomics and Bioinformatics,简称GPB)评选,成功入选2019年度“中国生物信息学十大进展”,该成果所用算法/工具入选2019年度“中国生物信息学十大算法和工具”。
随着三维基因组构象捕获实验技术(3C)尤其是与通量测序相结合技术(Hi-C)的发展,解析三维基因组结构成为了研究基因调控的常用手段。精确的三维基因组结构重构对于研究基因调控等生物过程和功能具有非常重要的意义。目前,绝大多数三维基因组结构重构算法均只是基于Hi-C数据。
曾坚阳研究团队将Hi-C和荧光原位杂交(FISH)数据整合起来,基于流形学习算法框架(GEM)提出了新的三维基因组结构建模方法—GEM-FISH。该方法采用一种分治策略,首先利用Hi-C和FISH数据构建拓扑关联结构域(TAD)之间的相对结构,再利用Hi-C数据构建TAD内部结构,最后将两者结合起来根据大分子能量性质优化微调,获得最终的结构。与现有方法相比,这一方法重构的基因组结构更为精确,平均相对误差更小,并且能够准确揭示出活跃状态和失活状态X染色体的差异。该方法被用来分析子结构分隔区在染色体三维空间的分布并得到了FISH实验数据的进一步验证。另外,该方法还可以用来分析超级增强子在染色体三维空间的分布,为基因调控研究提供更多的线索。
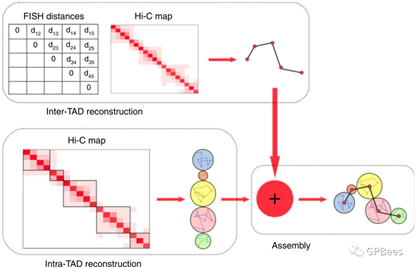
图:GEM-FISH通过整合Hi-C和FISH数据采用分治策略建模过程概览。
该研究成果第一作者是曾坚阳研究组已出站博士后Ahmed Abbas,通讯作者是曾坚阳副教授,合作者包括清华大学张奇伟教授和高军涛研究员等。研究成果得到国家自然科学基金等项目经费的支持。
“中国生物信息学十大进展”是由《基因组蛋白质组与生物信息学报》于2018年发起组织评选,该评选旨在推动我国生物信息学的学科发展和创新研究,充分展示和宣传我国生物信息学领域的重大研究成果,曾坚阳研究组三维基因组研究成果曾入选2018年度“中国生物信息学十大进展”。
论文原文链接:
https://www.nature.com/articles/s41467-019-10005-6
工具链接:
https://github.com/ahmedabbas81/GEM-FISH
(文/尚妤婵)