近日,清华大学交叉信息院曾坚阳研究组成功开发了从大规模科学文献中提取生物医学实体关系的深度学习模型,相关研究成果《A novel machine learning framework for automated biomedical relation extraction from large-scale literature repositories》于6月8日在《Nature Machine Intelligence》上在线发表。
理解药物、靶点、病毒、副作用等等生物医学实体之间的相互作用规律,是生物医学研究者们长期以来致力于探索和研究的问题,关于这些作用规律的研究成果广泛分布在超过3千万篇的科研文献当中,且文献的数量还在不断增加。目前,大多数知名的生物医学数据库,例如DrugBank、CTD、SIDER和BioGRID,都是由人类科学家花费大量的时间和精力从科学文献中整理而来的。虽然深度学习技术可以被用来加速这一过程,但在生物医学这种专业性领域,大规模的训练数据却并非能够轻易得到。为了解决这一问题,来自清华大学的曾坚阳研究团队采用了一种基于远监督的深度学习策略,使得模型能够在不依赖于人工标注数据的情况下应用到各种生物医学关系抽取场景当中。此外,作者所提出的集成了隐式句法树学习和注意力机制的模型,在多项生物医学关系抽取任务当中,都取得了领先的实验结果。这项研究成果表明,这种新型的机器学习框架能够为生物医学关系发现提供有力的帮助。目前,该工作已被应用到一项旨在从已有的老药中发现治疗COVID-19的潜在药物的工作当中,相关的研究成果已发布在生物预印本网站bioRxiv上。(https://www.biorxiv.org/content/10.1101/2020.03.11.986836v1)。
目前,曾坚阳研究组所提出的生物医学关系自动抽取框架已成功应用到多个生物医学场景当中,包括:
1. 通过抽取出的提示性信息指导了若干湿实验验证,从而确认了新的药物-靶点作用关系。
2. 在一项针对新冠肺炎的老药新用研发任务中,该关系抽取模型被应用到一个回顾性研究当中,即通过查找文献支持来验证针对SARS或MERS的老药新用策略的可行性,从而间接证明该老药新用策略针对COVID-19的有效性。
3. 针对更多的生物实体间的作用关系抽取,如病毒-宿主、药物-副作用间的关系抽取,该框架已在初步实验中验证了其有效性。
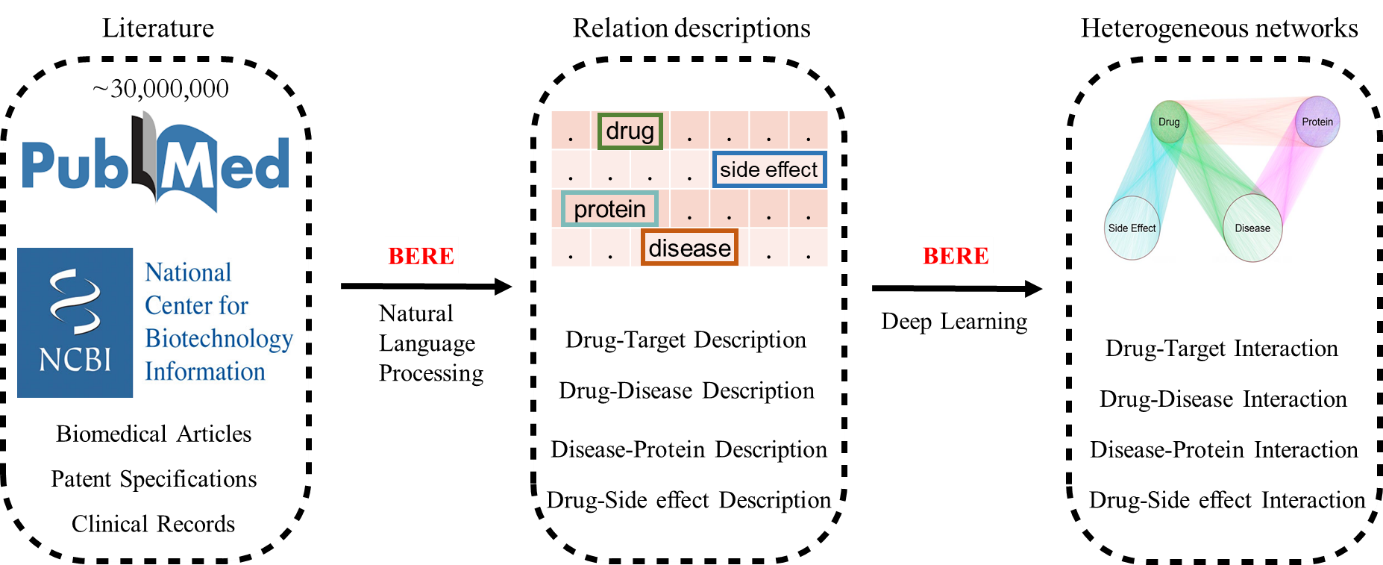 图:生物医学实体关系抽取的流程图
图:生物医学实体关系抽取的流程图
该论文通讯作者为清华大学交叉信息院曾坚阳副教授和赵诞助理研究员,第一作者为清华大学交叉信息研究院硕士生洪礼翔。该研究由国家自然科学基金、南京图灵人工智能研究院和中关村海华前沿信息技术研究院支持。
论文原文链接:https://www.nature.com/articles/s42256-020-0189-y