近日,清华大学交叉信息研究院的曾坚阳研究组成功开发了能在全基因组上预测RNA聚合酶暂停位点的深度神经网络模型,该研究为Pol II暂停现象在转录过程中的调控机制提供了一个全新的分析框架,也为在缺乏测序数据的细胞系上研究Pol II暂停提供了很好的预测参考模型。相关成果《基于机器学习框架的转录延伸建模》(A machine learning based framework for modeling transcription elongation) 于2月1日 在《美国国家科学院院刊》(Proceedings of the National Academy of Sciences of the United States of America)上发表。
真核生物的转录是一个高度复杂且被准确调控的动态过程,RNA聚合酶 II (RNA polymerase II, Pol II) 作为转录过程(特别是转录延伸阶段)的核心分子成员,对遗传信息的传递起着关键作用。研究表明,在转录延伸过程中,普遍存在Pol II暂停(Pol II pausing)现象,其参与下游基因的表达调控、影响基因可变剪切的发生,与细胞分化、发育等生命活动息息相关。目前,Pol II暂停相关的转录调控机制研究主要依赖于NET-seq(Native elongating transcript sequencing)等高通量测序技术,成本高、需要一定实验周期。因此,如何实现在全基因组水平上快速、高效的分析Pol II 暂停的偏好位点及其序列特征,以及其与相关转录因子间的关系,是转录调控领域亟待解决的技术难点。为了解决这一问题,来自清华大学的曾坚阳/赵诞团队采用基于注意力机制(attention mechanism)的卷积神经网络对全基因组上的Pol II暂停位点进行预测。在NET-seq数据集上,其预测准确率均显著优于现有机器学习模型。此外,研究组通过分析模型的注意力机制以及分析模型在基因组上特定位点的预测值,进一步探索了Pol II暂停的生物学特征以及和其他转录调控机制的关系,包括:
1. 发现了决定Pol II暂停的序列特征以及其与暂停位点的关系。
2. 通过分析不同可变剪切方式位点上Pol II的暂停倾向,提供了Pol II影响可变剪切的可能生物学机制。
3. 全面分析了Pol II与转录因子、组蛋白修饰、DNA甲基化发生的相关性。
4. 发现模型能够很好的弥补测序数据深度不足带来的缺陷。
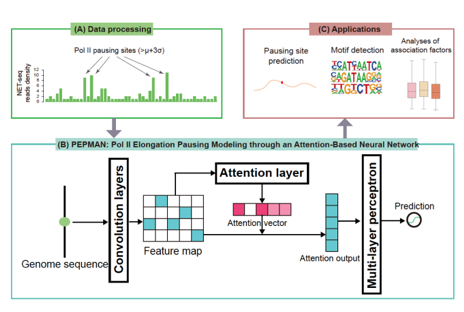
Pol II暂停分析预测框架图
该论文通讯作者为清华大学交叉信息研究院的曾坚阳副教授和赵诞助理研究员,第一作者为清华大学交叉信息研究院2016级博士研究生冯沛源和2016级硕士研究生肖安。此项研究工作获得国家自然科学基金、南京图灵人工智能研究院和中关村海华前沿信息技术研究院支持。
论文链接:https://www.pnas.org/content/118/6/e2007450118